研究人员创造了一个“罗塞塔石碑”来解码免疫识别

圣裘德儿童研究医院和弗雷德·哈钦森癌症研究中心的科学家们开发了一种算法,它的功能就像罗塞塔石碑,可以帮助破译免疫系统如何识别和结合抗原。这项研究今天在该杂志上作为提前在线出版物发表自然,应该有助于发展更个性化的癌症免疫疗法和先进的诊断和治疗传染病。
免疫系统依赖于一种叫做T的分子细胞受体在T的表面细胞识别和应答来自病毒感染细胞、肿瘤和其他威胁的外来抗原。基因组重排意味着大量不同的T细胞受体是有可能的。每个人都有大约1亿个不同的受体,被称为他们的T细胞库,即使在同卵双胞胎中也几乎没有重叠。库中的每个受体都能够识别不同的抗原,并召集免疫反应来应对这种威胁。
“到目前为止,这种惊人的多样性阻碍了对识别和响应相同抗原的T细胞受体进行分类的努力,”圣犹达免疫学系的副成员保罗·托马斯博士说。他和弗雷德·哈钦森癌症研究中心和西雅图华盛顿大学的菲利普·布拉德利博士是这项研究的共同通讯作者。
托马斯说:“这不仅阻碍了对免疫识别的理解,也阻碍了我们更有效地利用免疫系统来瞄准驱动肿瘤细胞的新病毒或突变的能力。”“通过这种算法,我们有一个系统来识别识别相同抗原的T细胞受体的关键特征以及它们如何相互作用。这为设计识别癌症或新病毒的受体奠定了基础。”
该算法是使用研究人员开发的工具来定义T细胞受体如何识别抗原的一部分,称为表位。表位显示在循环免疫细胞的表面,是T细胞结合抗原的地方免疫反应.多个抗原表位产生于相同的病毒、肿瘤或其他威胁。每个表位都被一群携带不同但特异性的T细胞受体的T细胞靶向识别和反应。
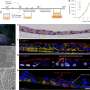
这些工具包括TCRdist,研究人员用它来计算T细胞受体关键特征的相似性和差异性,比如氨基酸序列在抗原识别的重要区域。TCRdist使科学家能够识别识别相同表位的T细胞受体。
“这些分析工具帮助我们以一种比以前更连贯的方式了解T细胞对特定抗原的反应。对给定表位的T细胞受体进行分组,揭示了大部分表位的潜在共同特征,”第一作者、托马斯实验室的工作人员、医学博士Pradyot Dash说。
就像学者们用来解码象形文字的罗塞塔石碑一样,研究人员用超过4600个T细胞受体来训练算法,然后用它来正确地将81%的人类T细胞和78%的小鼠T细胞分配到10个不同的病毒表位之一。“训练数据”来自78只感染了流感或巨细胞病毒(CMV)的小鼠和32名感染了流感、巨细胞病毒或爱泼斯坦-巴尔病毒的人类。每个T细胞的表位先前都是用一种不同的、更费力的方法确定的。
研究人员在不知道表位受体识别的情况下,在三只感染流感的老鼠身上测试了这种算法。该算法能够以高达90%的准确率预测这些细胞识别的流感表位。“事实上,85%的正确分类的T细胞受体以前没有被识别出来,”托马斯说。“这证明了这种方法对新型抗原特异性t细胞受体进行分类的能力。”
研究人员还注意到,表位包括具有关键相似性的T细胞受体簇以及不太相似的“异常”受体。托马斯推测,多达10%的T细胞受体是帮助免疫系统识别和快速响应突变的异常值,否则可能会帮助病毒感染的细胞和其他威胁延迟检测。
该项目始于2012年,当时以色列内盖夫的弗雷德·哈钦森和本-古里安大学的合著者托默·赫兹博士正在参观托马斯的实验室。托马斯说:“我们正在观察T细胞受体的氨基酸序列时,托默问了一个特定受体对应的表位是什么。”“如果你观察受体的时间足够长,你就能识别出氨基酸的模式。到那时,我们已经研究了很多受体,并很快确定它是一种流感PB1表位特异性受体。
“当时,没有好的计算工具来分析这些数据。托马斯说:“我们可以教计算机识别我们所做的相同模式,并利用这些信息来理解控制T细胞识别的规则。”“这开启了我们的合作。”
更多信息:Pradyot Dash等人,可量化的预测特征定义表位特异性T细胞受体谱,自然(2017)。DOI: 10.1038 / nature22383