癌症基因依赖图谱有助于揭示蛋白质之间的关系
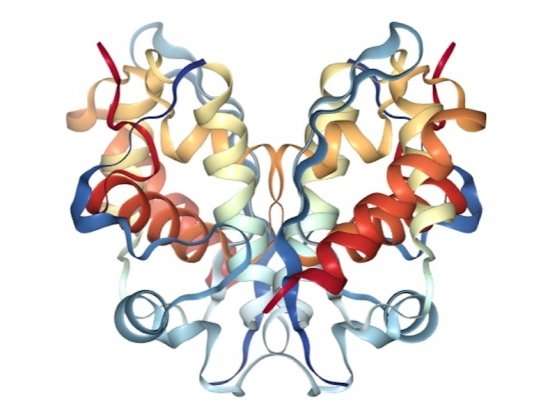
通过合并癌症功能基因数据与蛋白质相互作用的信息,科学家可以大规模探索蛋白质复合物。
“依赖性”图谱揭示了基因适应癌症细胞为了生存。虽然这种方法有助于为几种癌症类型确定有前途的治疗策略,但研究人员现在发现,它也提供了一个机会来探究癌症的复杂性蛋白质交互。
虽然测序工作可能已经确定了几乎所有与癌症有关的基因,但对这些基因的蛋白质产物以及它们如何相互作用的研究要困难得多。因为蛋白质共同工作来驱动大部分的细胞活动,所以大规模探索这些相互作用的能力可以提供对所有生物过程的洞察。
该报告发表在《细胞系统》(Cell Systems)杂志上,由研究生约书亚·潘(Joshua Pan)、计算生物学家罗宾·迈耶斯(Robin Meyers)、癌症数据科学组副主任Aviad Tsherniak和丹娜-法伯癌症研究所(Dana-Farber Cancer institute)成员、表观基因组学项目联席主任西格尔·卡多奇(Cigall Kadoch)领导的布罗德研究所团队,描述了他们如何将来自癌症依赖图谱(DepMap)项目的基因组级依赖数据与现有的大型蛋白质相互作用数据集相结合,创建了一个用于检查蛋白质复合体(执行协调任务,如基因转录的蛋白质组装)的框架。
该团队的基本假设是:
- 当一个基因被敲除(用CRISPR)或沉默(用RNA干扰)时,细胞就不能再制造该基因的蛋白质产物。结果,细胞的功能状态或生存(即“适应性”)发生了变化。
- 两种失去的蛋白质具有相似的适应度影响,它们可能具有相似的功能,甚至可能是相同的成员蛋白质复合体。
- 通过在聚合屏幕上分析所有这些“敲除”如何影响细胞健康,并在数百个癌细胞系中比较这些“敲除”,研究人员可以根据功能和相互作用对蛋白质进行分组,探索已知蛋白质复合物成员的作用,并突出以前未被识别的蛋白质。
DepMap数据集包括了342个癌细胞系的CRISPR数据和501个RNAi数据,提供了一个完美的起点。将这些数据与最近七个蛋白质相互作用数据集进行整合,该团队开发了计算机模型,根据它们对细胞适应性的影响,对数百种人类蛋白质复合物的成分进行了评分和分组。这些健康分组符合蛋白质的功能和生化关系。
以研究充分的复合物为基准,该团队的模型准确地捕获了已知的蛋白质组成、模块层次,甚至结构关系的组成列表。该模型还指出了不同的、功能不同的复合物之间共享的亚基,并确定了一个以前未被识别的复合物的组成部分,即SWI/SNF或BAF(其成员在癌症中经常发生突变)。
研究人员认为,随着新的适应度或来自其他细胞系的依赖数据的获得,他们的模型只会增强其威力,并有助于阐明与疾病相关的基因变异或结构变化对蛋白质相互作用和疾病生物学的影响。
进一步探索






用户评论