新的多管齐下的方法探测非编码DNA如何影响基因表达

大约98%的人类基因组由非编码DNA组成,包括增强子、启动子和其他调节基因活性的元素。研究这些区域的方法往往是昂贵的、劳动密集型的,而且基本上是低通量的。
然而,为了真正理解非编码基因组的功能地理,研究人员需要一种方法来同时、快速、精确地分离和表征其中数千到数百万个调控DNA元件高分辨率.这种需求是巨大的,因为在性状和疾病的全基因组关联研究中发现的90%以上的变异都位于非编码DNA中。
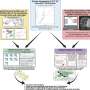
通过结合两种强大的基于测序的分析机器学习的工具,a研究小组由王新晨(音译)和副成员Melina Claussnitzer和Manolis Kellis领导的布罗德研究所代谢和表观基因组学项目设计了一种强大的新方法,用于测量单个非编码DNA片段的控制能力基因表达,并且在大范围和高分辨率下都能做到。
这种方法被称为HiDRA(调节活动的高分辨率解剖),它汇集了一些广泛使用的工具:
- ATAC-seq(高通量测序转座酶-可接近染色质分析),是Broad公司副成员Jason Buenrostro开发的一种技术整个基因组用于解开的DNA可转录区域;
- STARR-seq(自我转录活性调控区测序),由Kellis实验室alum Alexander Stark开发的用于测量非编码DNA片段表达促进活性的测定方法;而且
- SHARPR- re是一种基于SHARPR工具的机器学习算法,Kellis的实验室开发用于分析大规模并行报告分析的数据。
在这些方法的基础上,HiDRA允许研究人员创建大量的调控DNA文库,并以核苷酸水平的分辨率研究它们对基因表达的影响。
正如他们在自然通讯该团队将HiDRA应用于一个血细胞系,测试了约700万个非编码DNA片段,以寻找调节基因表达的片段,最终确定了6.5万个。这些片段包括明显标记为增强子和启动子的片段,以及缺乏这种标记的片段,这表明基因组可能包含我们尚未发现的其他类型的表达控制元件。
此外,该团队使用HiDRA来检查与不增加风险的变体相比,调控元件中的疾病风险DNA变体如何影响基因表达——这对于寻求研究启动子和增强子中的微小序列变化如何影响人类特征和疾病状态的研究人员来说是一个好处。
该团队的研究结果表明,HiDRA是一种可推广的方法,用于剖析基因调控的细微差别和各种功能元素在人类疾病中所起的作用。
更多信息:王欣晨等。人类转录调节区域和核苷酸的高分辨率全基因组功能解剖,自然通讯(2018)。DOI: 10.1038 / s41467 - 018 - 07746 - 1