有助于在人类基因组中找到删除和重复的新方法
一种新的机器学习方法准确地识别出人类基因组中被复制或删除的区域——称为拷贝数变异——这通常与自闭症和其他神经发育障碍有关。这种新方法是由宾夕法尼亚州立大学的研究人员开发的,它整合了几种算法的数据,这些算法试图从外显子组测序数据中识别拷贝数变异——仅对人类基因组蛋白质编码区域进行高通量DNA测序。一篇描述该方法的论文,它可以帮助临床医生提供更准确的诊断基因疾病,出现在7月的期刊杂志基因组研究。
“外显子组测序正迅速成为临床环境中识别基因变异的金标准,因为它比其他方法更快、更便宜,”宾夕法尼亚州立大学生物化学和分子生物学副教授、该论文的主要作者Santhosh Girirajan说。然而,目前从外显子组测序数据中识别拷贝数变异的算法存在非常高的假阳性率——他们识别的许多变异实际上不是真实的。通过我们的新方法,称为“CN-Learn”,我们报告的拷贝数变异大约90%是真实的。
人类基因组通常包含每个基因的两个副本,染色体对的每个成员都有一个副本。当一个细胞分裂成两个时,基因组就会被复制,使得每个子细胞得到一个完整的补充基因但在基因组复制过程中偶尔会出现错误,这些错误出现在精子或卵细胞中,可能导致个体获得多于或少于两个基因副本。
为了从外显子组测序数据中识别拷贝数变异,研究人员观察了每个基因产生的DNA序列的相对数量。如果一个个体中只有一个基因的拷贝,他们期望看到的测序读取量比有两个拷贝时要少,而一个基因的三个拷贝会导致更多的读取量。但这并没有那么简单,因为许多其他因素可以影响每个基因产生多少测序reads。因此,研究人员开发了几种算法来尝试正确识别复制数字变体从外显子组测序数据。然而,单独来看,这些算法并不是特别可靠。
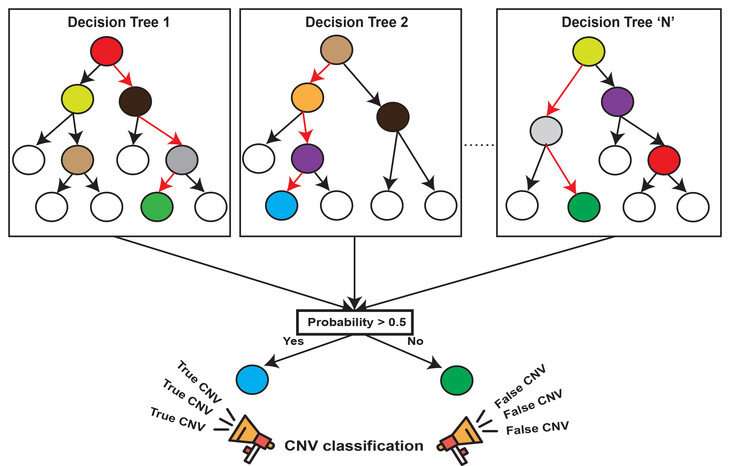
“通常,通过使用多种算法并仅计算由多种算法计算的副本 - 变体算法的大量误报,并仅计算由所有方法鉴定的venn图所识别的变体,”研究生vijaykumar pounraja说宾夕法尼亚州的第一个作者。“这种方法具有多种缺点和局限性,因此我们决定制定新的机器学习方法。”
CN-GROEWS集成来自四种不同副本的数据 -数量-Variant算法,并使用一小部分生物学验证的删除和重复来学习这些基因组事件的签名。这学习过程通过机器学习促进算法被称为随机森林,它使用数百人决策树建立缺失和重复的遗传背景与验证它们的可能性之间的关系模型。然后,CN-Learn使用这个模型来预测没有验证的其他样本中的删除和重复。
“关于患者诊断和最终治疗的决定是根据这些信息制定的,因此让他们对此非常重要,”格里拉泽说。“因为这一点,我们制作了CN-GROUSE和所有必要的支持程序可在一个简单的包装中下载。”
进一步探索











用户评论