深度学习有助于预测对抗COVID-19的新药物组合
COVID-19的存在威胁突出表明,迫切需要针对新出现的健康威胁开发有效的治疗方法。深度学习给我们带来的奢侈品之一是,我们能够在事态发展的过程中调整形势——只要我们能跟上病毒式传播的威胁,并获得正确的数据。
与所有新的医学疾病一样,数据往往需要时间才能跟上,而病毒很快就会减速,这是一个艰巨的挑战,因为它可以迅速变异,并对现有药物产生耐药性。这让麻省理工学院计算机科学和人工智能实验室(CSAIL)的科学家们提出了一个问题:我们如何才能确定针对快速传播的SARS-CoV-2的正确协同药物组合?
通常,数据科学家使用深度学习根据现有的大量数据集挑选出药物组合,比如癌症和心血管病但可以理解的是,它们不能用于数据有限的新疾病。
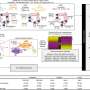
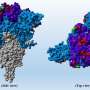
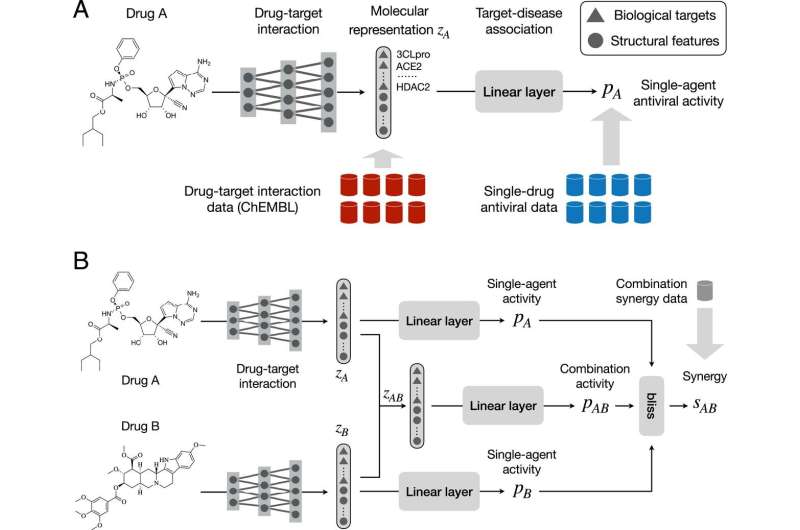
在没有必要的事实和数据的情况下,该团队需要一种新的方法:一种双重性的神经网络。由于药物协同作用通常是通过抑制生物靶点(如蛋白质或核酸)发生的,因此模型联合学习药物-靶点相互作用和药物-药物协同作用来挖掘新的组合。药物靶点预测器对药物与一组与所选疾病相关的已知生物靶点之间的相互作用进行建模。目标疾病关联预测器学会了解药物的抗病毒活性,这意味着确定病毒在受感染组织培养中的产量。它们可以一起预测两种药物的协同作用。
两个新药物组合发现了瑞德西韦(目前已获FDA批准用于治疗COVID-19)、利血平以及瑞德西韦和IQ-1S,这两种药物在生物试验中被证明对该病毒具有强大的抑制作用。
“通过建模药物和生物靶点之间的相互作用,我们可以显著减少对组合协同数据的依赖,”CSAIL博士、麻省理工学院布罗德研究所博士后金文公(Wengong Jin)说,他是一篇关于该研究的新论文的第一作者。“与以前使用药物-靶点相互作用作为固定描述符的方法相比,我们的方法学会了从分子结构预测药物-靶点相互作用。这是有利的,因为大部分化合物具有不完全的药物-靶点相互作用信息。”
使用多种药物以最大限度地提高药效,同时减少副作用,对于上述癌症和心血管疾病,包括许多其他疾病,如结核病、麻风病、疟疾,这种方法几乎无处不在。非常重要的是,使用专门的混合药物可以减少严重的、有时是公开的耐药性威胁(想想耐甲氧西林金黄色葡萄球菌,又称“MRSA”),因为许多耐药突变是相互排斥的。对一个病毒来说,同时产生两种突变,然后在联合治疗中对两种药物产生耐药性,这要难得多。
该模型也不仅仅局限于sars - cov -2,它也可以用于传染性越来越强的delta变体。要扩展到那里,你只需要额外的药物组合协同作用的突变数据。该团队还将他们的方法应用于艾滋病毒和胰腺癌。
为了进一步完善他们的生物模型,该团队计划加入更多的信息,如蛋白质-蛋白质相互作用和基因调节网络。
他们正在探索的另一个未来工作方向是所谓的“主动学习”。许多药物由于组合模型的尺寸有限,它们倾向于某些化学空间,因此在预测中存在很高的不确定性。主动学习有助于指导数据收集过程,并在更广阔的化学空间中提高准确性。
这项研究发表在美国国家科学院院刊。