利用基因组学来匹配治疗,改善了某些转移性乳腺癌患者的预后

根据ESMO分子靶点临床可动性分级(ESCAT)的I/II层,使用多基因测序作为治疗决策工具改善了转移性乳腺癌患者的预后SAFIR02-BREAST审判在圣安东尼奥乳腺癌研讨会,于2021年12月7日至10日举行。
下一代基因组分析技术允许研究人员同时对多个基因进行测序,并建立患者肿瘤的突变谱。这有助于针对可能不是该疾病的标准护理的治疗方法所确定的改变,以改善患者预后为目标。
“多基因测序已被广泛应用,但其临床影响和最佳使用框架尚不清楚,”Gustave Roussy癌症校区的研究主任、医学博士Fabrice André说。“我们研究的主要目的是测试基因组分析是否对转移性乳腺癌患者有用,以及我们如何才能最好地分析结果。”
二期SAFIR02-BREAST试验招募了转移性her2阴性乳腺癌患者,以评估基因组学指导下的靶向治疗与维持化疗相比是否能改善无进展生存期(PFS)。接受过两种以上化疗或试验中评估的一种靶向治疗的患者不符合参加试验的资格。
研究人员对该试验进行了综合分析SAFIR-PI3K试验该研究比较了pi3k α特异性抑制剂alpelisib (Piqray)和雌激素受体拮抗剂fulvestrant与pik3ca突变转移性乳腺癌患者的维持化疗联合使用。
通过基因组分析新一代测序和SNP阵列对1462例患者进行了检测。研究人员分配了238名患者,这些患者在6到8个周期的化疗后病情稳定,并且对与其基因组改变(157例)匹配的适当靶向治疗(81例)进行了已知的基因组改变。
本研究纳入的药物为vistusertib、AZD4547、capivasertib、sapitinib、selumetinib (Koselugo)、vandetanib (Caprelsa)、bicalutamide (Casodex)、olaparib (Lynparza)和alpelisib,分别与以下靶点匹配:m-TOR、EGFR、AKT、HER2或EGFR、MEK、VEGF或EGFR、雄激素受体和PARP。
对患者肿瘤的基因组改变进行分类ESCAT量表,它对基因组改变作为治疗靶点的可能性进行了排名,基于来自临床研究.研究人员评估了与ESCAT排名相关的治疗效果。
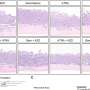
在115例ESCAT I/II基因组改变患者中,在匹配的靶向治疗和维持化疗组中,中位PFS分别为9.1和2.8个月。相反,在总体人群中,两组之间的PFS没有显著差异,当匹配到未达到ESCAT I/II级的改变时,靶向治疗是无效的,这表明ESCAT分类对匹配到基因组改变的靶向治疗的收益具有高度的预测性。
“我们的研究表明,基因组分析改善了癌症患者的预后转移性乳腺癌如果他们进行了ESCAT I/II类的修改,”André评论道。“这些发现表明,基因组学应该是护理途径的一部分,但如果没有使用确定的基因改变的可操作性的经过验证的框架来解释结果,它就没有影响。”
除了检测已知的基因改变,多基因测序还能让研究人员发现新的基因改变。André和同事们发现了21个与转移性进化相关的基因扩增或缺失,预后不良、耐药性或敏感性。
André补充说:“我们研究的总体含义是,如果使用正确的工具,精确医疗可以改善患者的预后。”
根据作者的说法,这项研究的主要局限性是靶向疗法的数量有限基因组的改变发现。