研究人员开发了一种用于分析多路组织成像数据的新软件
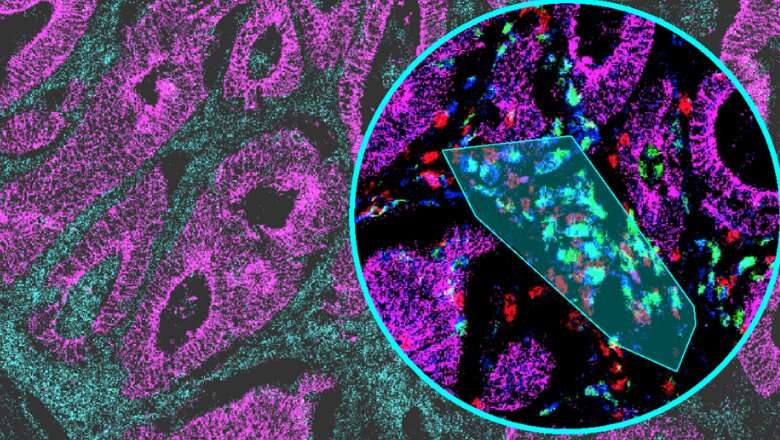
由Francesca Ciccarelli教授领导的伦敦国王学院癌症与药物科学学院、免疫学和微生物科学学院以及弗朗西斯克里克研究所癌症系统生物学实验室的一组研究人员发表了一篇新论文,展示了用于分析复杂组织成像数据的新软件。
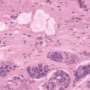
最近开发的多重成像技术,如成像大规模细胞术(IMC),能够详细研究组织细胞组成,这对于绘制疾病的组织水平特征和预测依赖于组织环境的治疗(如癌症免疫治疗)的结果具有特殊价值。这些技术通常会产生大量难以分析的高维数据,通常需要多个工具和具有计算专业知识的用户。
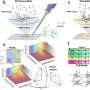
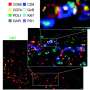
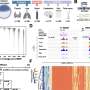
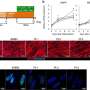
SIMPLI(从多路复用图像中进行单细胞识别)是一种灵活的识别方法软件这统一了多路成像数据分析的所有步骤,提供了一种改进的高维替代方案图像数据非专业人士也可以进行分析。SIMPLI对每个组织切片进行空间分辨率的单细胞分析,以及标记物表达的细胞独立量化,以研究在细胞水平上无法检测到的特征。它还生成表格和图形输出,便于在分析的每个步骤中对结果进行可视化检查。
“几年前,当我们开始处理高维IMC数据时,没有太多的分析工具。开发我们自己的工具非常有趣,这要感谢CRUK KHP中心资助的博士生Michele Bortolomeazzi的才华,并利用了Jo Spencer教授在组织组织学方面的广泛专业知识,”Francesca Ciccarelli教授说。
为了测试SIMPLI的性能和通用性,研究人员分析了使用不同成像技术(包括但不限于IMC)获得的图像,这些图像在来源、大小和分辨率上都是不同的。
伦敦国王学院是欧洲首批拥有Helios成像细胞仪的机构之一,该仪器可在BRC流式细胞仪平台上使用。
更多信息:Michele Bortolomeazzi等人,在单细胞分辨率下进行空间分辨组织表型的SIMPLI(从多路复用图像中识别单细胞)方法,自然通讯(2022)。DOI: 10.1038 / s41467 - 022 - 28470 - x