精制微生物“指纹”方法跟踪单个肠道微生物菌株的变异
凯西·d·莫罗博士和他的同事们在伯明翰阿拉巴马大学以前开发的一个叫WSS的微生物组“指纹”方法,识别单一特定的肠道细菌菌株,通过宏基因组数据的分析从粪便样本。他们表明,特定的菌株在成年人倾向于保持稳定一段时间后,除非摄动的事件(如抗生素或肥胖手术。他们还发现捐赠粪便移植应变给治疗梭状芽胞杆菌耐药感染坚持收件人只要移植后两年。
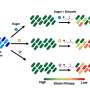
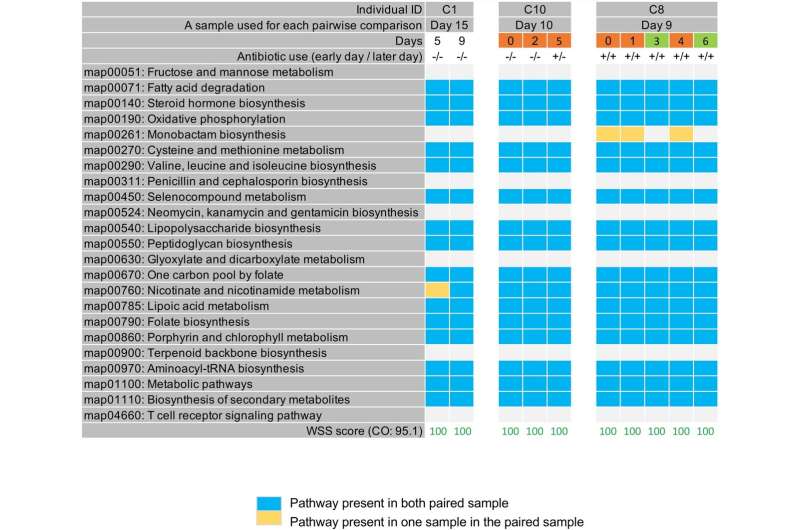
明天和Hyunmin古博士细化指纹方法寻找单核苷酸变异KEGG代谢途径的一个特定的菌株。这些变体可以识别单个应变被WSS的菌子。看菌子的拟杆菌vulgatus压力,例如,明日和古检查23个KEGG代谢途径出现在细菌。
他们现在这放大分析应用于监控菌子的变化在较短的一段时间,几天或几周,在两个关键肠道bacteria-B。vulgatus和拟杆菌均匀化。比较少量的健康个体和COVID-19住院病人,他们看到一个变种动力学的差异,他们说放缓预示着一个内在的应变率变化的病人。这种放缓可能最终导致微生物菌株社区的失调可能预示着改变肠道微生物的主要菌株。
拟杆菌属的物种的丰度高肠道菌群,他们可能关键物种,生物帮助定义一个完整的生态系统。
古永锵和明日的研究,“早期的微生物指标压力失调在人类肠道微生物群落的健康人和COVID - 19住院病人,”发表在《华尔街日报》科学报告。
古永锵和明日第一分析了以前公布的宏基因组数据从41个人采样相隔一年和11个人采样90天。他们看着一个占主导地位的b vulgatus在每个单独的两个时间点,他们不知道是否有显示不同的代谢变种KEGG模式,从分析检测单核苷酸变异在KEGG代谢途径,繁荣正义党。一般来说,大多数显示不同变种PKS模式这两个时间点之间的每一个人。
UAB研究人员分析了之前发布的宏基因组数据从六个健康个体采样每隔几天在3到10周,再次分析单核苷酸变异在23个KEGG菌子代谢途径。三人表现出不同的变种在每一个时间点,而三个显示菌子PKS模式出现了,消失了,再次出现在不同的时间点。
与其共享模式中也看到两三个COVID-19住院患者多次抽样。
“我们表明,肠道微生物群落在压力之下,例如那些COVID-19住院病人,可能状态指示的潜在转移占主导地位的应变会淘汰一个小应变,“古说。“破坏肠道微生物群落变化可能造成的压力,进而改变群落结构和功能代谢和殖民影响阻力。”
“一个复杂的功能之一生物系统是,当它接近一个关键的过渡,有减缓内在的改变,”明天说。“系统进入一个与自相关的条件,在时间点之间的模式将会重复。可能共享KEGG代谢途径集群代表自相关的状态在肠道微生物菌株社区,预示着一个应变的变化。”