身体如何识别有用的细菌
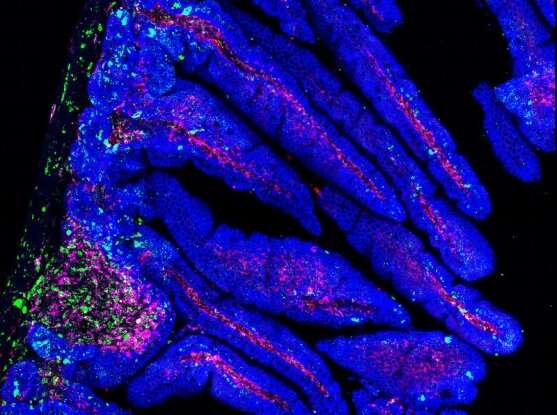
数万亿个细菌和其他微生物在我们的肠道微生物中蓬勃发展,在过去的十年中,科学家已经越来越意识到他们在创造健康的胃肠道环境中所起的至关重要的作用。整合到多种微生物环境中,是数百个负责避开外国入侵者的免疫系统细胞,使科学家受到一个重要问题的困扰 - 人体的自然防御能力如何告诉有用和有害的细菌?
在一项发表的研究中免疫,麻省理工学院和哈佛大学研究所的传染病和微生物组计划(IDMP)的研究人员以及马萨诸塞州综合医院的计算和综合生物学中心以及分子生物学系迈出了回答该问题的新一步。用一个计算算法他们开发的是在微生物基因中寻找抗原形成的DNA序列,他们发现了通过免疫系统识别的几种抗原,但可以忍受。
在t中发现了一种抗原,SUSC,并耐受细胞团队观察到的每个健康人。但是,在克罗恩病的患者中炎症性肠病这影响了美国超过一百万的人 - 对SUSC的T细胞反应看起来截然不同。研究人员认为,有一天可以使用SUSC和类似抗原来监测疾病,并为患有免疫相关肠道疾病的患者提出治疗方法。
“对于在克罗恩氏病中发生耀斑的人们,他们的T细胞认识到这一点微生物组抗原,但没有表现出对它的宽容,而是有一个炎症反应这项研究的合着者兼IDMP功能基因组学总监丹尼尔·格雷厄姆(Daniel Graham)说:“这表明我们可以使用这些反应来监测患者的疾病。”
在T细胞的眼中
在实验之前,格雷厄姆(Graham),第一作者兼访问研究生托马斯·佩德森是为什么为什么免疫细胞不要攻击有益的肠道微生物。目前尚不清楚鉴定体内外源病原体的助手T细胞是否正在积极识别和耐受微生物抗原,或者只是完全对它们视而不见。第三个理论表明,定期接触抗原可能会定期驱动人体最终会习惯的微小免疫反应。缺乏支持任何理论的数据使研究人员陷入黑暗。
格雷厄姆解释说:“不知道T细胞看到什么,您无法回答这些问题。”
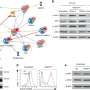
为了了解T细胞的观点,该团队获得了他们在2018年构建的计算机程序的帮助,称为细菌原始T细胞抗原(BOTA)预测指标。BOTA最初开发了用于研究小鼠的,允许科学家筛选大量细菌基因组序列数据,以预测哪些基因负责编码不同的微生物抗原。通过对BOTA算法进行了几次更改,使其适用于人类(他们命名为HBOTA),该团队开始在人类肠道微生物组中寻找常见的抗原。
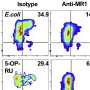
HBOTA分析表明,超过60种常见抗原,主要由不同个体的微生物组成的小细菌片段组成。该小组发现,健康参与者的T细胞积极识别和耐受这些片段而没有安装炎症反应,这肯定的迹象表明抗原在调整受试者的T细胞活性方面发挥了一定作用。
研究人员希望找到“通用”肠道微生物组抗原,这些抗原受到许多人(如果不是全部)人类参与者的T细胞所耐受的耐受性,使SUSC的发现更加令人兴奋。他们确定抗原是一种在细菌细胞中运输营养的复合物的一部分,表明SUSC对于许多微生物的生存和生长至关重要,并且其在肠道微生物组中的丰度本身可能是其耐受性的关键。
“多年来,Broad Institute的微生物组计划和克罗恩病的努力已经从识别启动剂微生物组中的幼稚CD患者中脱颖而出,阐明了微生物如何教育免疫系统,将特定的细菌代谢物与炎症联系起来,现在提出了蓝图以识别蓝图以识别微生物的表现,以识别微生物的表现。或激活T细胞,” Xavier说。
临床可能性
SUSC的发现使团队有能力超越其原始实验,并探索来自胃肠道疾病患者的T细胞如何对肠道细菌的反应。他们发现,来自克罗恩病患者的T细胞识别出SUSC,并安装了相当大的免疫反应,标志着高产生IL-17(一种促炎性细胞因子)。相比之下,来自健康患者的T细胞通过产生IL-10对SUSC做出反应,IL-10具有明显的抗炎作用。
该小组认为,有可能使用SUSC和类似的抗原来监测克罗恩病的进展,从而有可能使临床医生在疾病发生之前预测疾病。他们现在正在努力更好地了解背后的机制免疫系统对细菌抗原的反应,这可能有一天会导致克罗恩病和其他炎症性肠道疾病的治疗。
格雷厄姆说:“我们在这项研究中表明,T细胞对微生物组的反应非常动态。”“如果我们能够理解这些过程的基础机制,也许我们可以设计有针对性的治疗剂来干预和保持对微生物组的耐受性。”
进一步探索
Dirk Gevers等人,新发病的克罗恩病的治疗微生物组,细胞宿主和微生物(2014)。doi:10.1016/j.chom.2014.02.005
Tommi Vatanen等人,微生物组LPS免疫原性的变化有助于人类自身免疫性,细胞(2016)。doi:10.1016/j.cell.2016.04.007
Matthew T. Henke等人,Ruminococcus Gnavus是与克罗恩病相关的人类肠道微生物组成员,它产生了炎症多糖,美国国家科学院论文集(2019)。doi:10.1073/pnas.1904099116