基因相互作用分类肠肿瘤为个性化医疗
根据今天发表在《柳叶刀》杂志上的一项研究,基于肿瘤细胞内常见的基因相互作用模式,肠癌可以分为六种具有临床意义的亚型eLife.
这种新的肠癌分类方法将有助于对这种疾病的理解,并最终可能被医生用于精准医疗——帮助他们为个别患者量身定制最佳治疗方案。
“目前,疾病分期被用于指导肠癌患者的临床管理,但这是不够的,因为在相似阶段的肿瘤之间可能存在实质性差异,”该研究的第一作者、中国郑州大学第一附属医院的卢早渠博士解释说。
“更详细的肠癌分子分类更有效,但它仍然是基于肿瘤内某个时间点的基因活性水平的快照(例如,在活组织检查时),并不能反映基因活性的变化动态以及如何变化基因相互交流。”
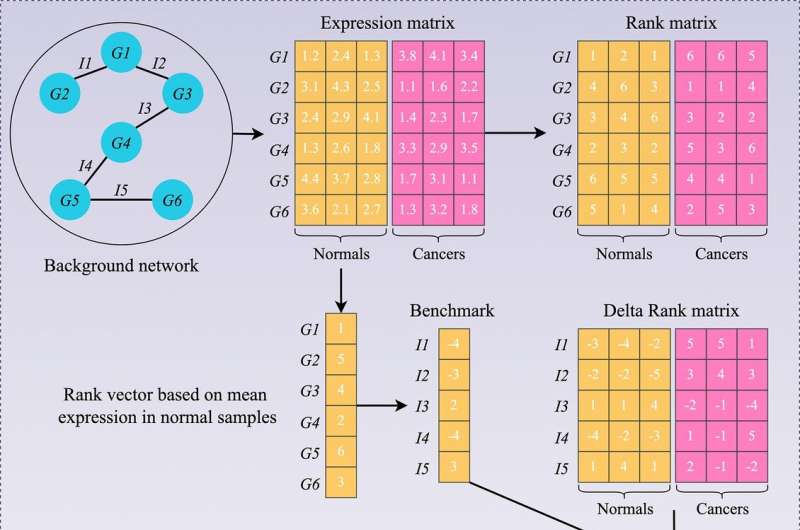
相比之下,生物相互作用网络——即基因之间通常相互作用的图谱——无论何时都保持相对稳定,并且可以更可靠地表征组织。此外,基因相互作用在正常情况下是高度保守的组织但在疾病中发生了广泛的改变。通过量化不同基因对的“相互作用变化”,可以将病变组织中基因相互作用的总体变化与正常组织中的背景水平进行比较。
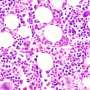
该团队应用了这种基因相互作用网络(GIN)方法,使用了2000多个肠癌组织样本和308个正常肠道组织样本,构建了基因相互作用变化的大规模网络。利用该网络,他们能够将肠癌分为六种亚型,每种亚型具有不同的临床和分子特征。这些特征包括重要的肿瘤特征,如癌细胞与其他细胞的比例细胞类型,以及癌症细胞与干细胞类似,干细胞可以影响肿瘤生长和扩散的可能性。
GIN分类还确定了具有不同免疫原性的组,即肠肿瘤组织刺激肿瘤的可能性有多大免疫反应这对于确定患者是否对免疫疗法有反应至关重要。其他有用的特征包括可能的预后,对放射治疗和免疫治疗的耐药性,以及对不同化疗药物的敏感性-所有这些都可以帮助定制治疗。当GIN方法在另外19个肠癌数据集中进行测试时,同样的6种亚型被一致识别出来。
第一个GIN基于近1400个基因之间的2000多个相互作用,并基于289个基因产生了一个分类器。这是不可能测试的临床因此,为了提供一种快速可用的临床工具,该团队将分类器简化为随机选择的14个基因迷你分类器。当他们在214个肠癌样本的子集上使用迷你分类器时,他们发现在识别六种肠癌亚型时,它与完整分类器一样稳定。
“我们的研究已经确定并验证了一个高分辨率的分类系统,它可以作为优化肠癌患者治疗决策的工具,”中国郑州大学第一附属医院介入放射科教授兼主任、资深作者韩信伟说。
“下一步将在前瞻性临床试验中确认六种肠癌亚型的生物学和临床可解释性,但我们相信这种新的分类法可以促进对患有肠癌的患者进行更有效的个性化治疗肠癌."