科学家开发了强大的人工智能算法,以获得对致命胶质母细胞瘤的新见解

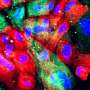
迈阿密大学米勒医学院西尔维斯特综合癌症中心的科学家们与国际研究人员合作,开发了一种复杂的AI算法,可以执行先进的计算分析,以确定多形性胶质母细胞瘤(GBM)和其他癌症的潜在治疗靶点。
他们的研究发表在2月2日的杂志上自然癌症这可能对GBM(一种侵袭性的、通常致命的脑癌)以及某些乳腺癌、肺癌和儿科癌症的未来治疗产生深远影响。
西尔维斯特综合癌症中心副主任、该研究的资深作者Antonio Iavarone医学博士解释说:“我们的工作代表了转化科学,为改变胶质母细胞瘤患者在临床的常规管理方式提供了直接的机会。”“我们的算法为精准癌症医学提供了应用,为肿瘤学家提供了对抗这种致命疾病和其他癌症的新工具。”
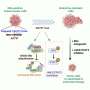
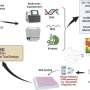
这种AI算法被称为sphinks -基于基板磷酸位点的激酶网络推理,它部署了深度机器学习来帮助研究人员识别和实验验证两种方法蛋白激酶(PKCδ和DNAPKcs)作为相关的罪魁祸首肿瘤进展两种GBM亚型和as潜在的治疗靶点对于其他癌症。
蛋白激酶是目前用于精准癌症医学的关键靶点,用于针对患者的特定癌症特性进行定制治疗。研究人员在论文中将最活跃的激酶称为“主激酶”,这些激酶是临床医生将靶向药物作为当前癌症治疗的标志之一。
除了识别主激酶,伊瓦隆博士和同事还使用了肿瘤在实验室中从患者样本中培养的类器官——他们称之为“患者衍生的肿瘤化身”——表明干扰主激酶活性的靶向药物可以阻止肿瘤生长。
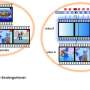
此前,Iavarone博士和他的团队报告了一种新的胶质母细胞瘤分类通过捕捉关键的肿瘤细胞特征,并根据其生存可能性和其预后对GBM患者进行分组肿瘤对药物的易感性.在这项新研究中,这些分类是通过几个组学平台独立确认的:基因组学(基因)、蛋白质组学(蛋白质)、脂质组学(脂肪分子)、乙酰组学(表观遗传学)、代谢组学(代谢物)和其他。
SPHINKS利用机器学习来完善这些组学数据集,并创建一个相互作用组-一套完整的生物相互作用-以精确定位每种胶质母细胞瘤亚型中产生异常生长和治疗耐药性的激酶。这些发现表明,多组学数据可以生成新的算法,根据每个患者的胶质母细胞瘤亚型,预测哪些靶向疗法可以提供最佳的治疗方案。
“我们现在可以根据不同组学之间共同的生物学特征对胶质母细胞瘤患者进行分层,”伊瓦隆博士说。“仅仅阅读基因组是不够的。我们需要更全面的数据来确定肿瘤的脆弱性。”
尽管许多其他癌症都取得了突破性进展,但胶质母细胞瘤患者的预后却十分惨淡——5年生存率低于10%。虽然许多药物正在开发作为潜在的治疗,临床医生需要一种方法来识别分子机制驱动每个病人的疾病,并适用于精准癌症医学。
根据研究人员的说法,SPHINKS算法和相关方法可以很容易地纳入分子病理学实验室。他们的论文包括一个临床分类器,可以帮助为每个患者分配适当的胶质母细胞瘤亚型。该团队还建立了一个门户网站访问算法。作者认为,这种方法可以产生有深刻见解的信息,可使多达75%的胶质母细胞瘤患者受益。
“这个分类器基本上可以在任何实验室使用,”安娜·拉索雷拉医学博士说,她是西尔维斯特CCC生物化学和分子生物学教授,也是这项研究的共同高级作者。“通过将组学信息导入门户网站,病理学家可以收到一个肿瘤、十个肿瘤的分类信息,无论他们导入多少个。这些分类可以立即应用于患者护理。”
虽然SPHINKS首次在胶质母细胞瘤上进行了测试,但该算法同样适用于其他几种癌症。研究小组在乳腺癌、肺癌和小儿脑瘤中发现了同样的致癌激酶。Drs。Iavarone和Lasorella及其同事认为,这一发现可能是一种新型临床试验的动力。
“我们正在探索一揽子试验的概念,”伊瓦隆博士解释说,“这将包括具有相同生物亚型但不一定是相同癌症类型的患者。如果患者患有胶质母细胞瘤或乳房或肺部癌症具有相似的分子特征,它们可能被纳入同一试验,”他继续说。“与其对一种药物进行多次试验,我们可以进行一次联合试验,并可能更快地为更多患者提供更有效的药物。”
更多信息:Anna Lasorella,综合多组学网络识别PKCδ和DNA-PK作为胶质母细胞瘤亚型的主激酶,并指导靶向癌症治疗,自然癌症(2023)。DOI: 10.1038 / s43018 - 022 - 00510 - x.www.nature.com/articles/s43018 - 022 - 00510 - x